Operaciones básicas de entrada/salida de archivos con Uproot#
¿Qué es Uproot?#
Uproot es un paquete de Python que lee y escribe archivos ROOT, y está únicamente enfocado en la lectura y escritura (sin análisis, sin gráficos, etc.). Interactúa con NumPy, Awkward Array y Pandas para cálculos, boost-histogram/hist para manipulación y visualización de histogramas, Vector para funciones y transformaciones de vectores de Lorentz, Coffea para escalado, etc.
Uproot está implementado solo con Python y librerías de Python. No tiene una parte compilada ni requiere una versión específica de ROOT. (Esto significa que si usas ROOT para algo más que entrada/salida, tu elección de versión de ROOT no estará limitada por la entrada/salida).
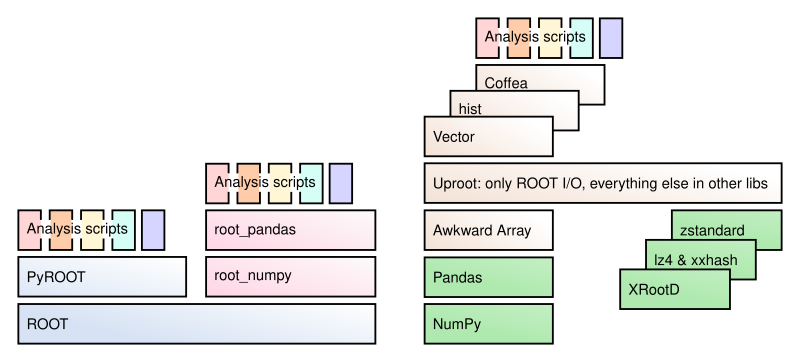
Como consecuencia de ser una implementación independiente de entrata/salida de ROOT, Uproot podría no ser capaz de leer/escribir ciertos tipos de datos. Qué tipos de datos no están implementados es un objetivo en constante movimiento, ya que siempre se están agregando nuevos. Una buena forma de leer datos es simplemente intentarlo y ver si Uproot genera algún error. Para la escritura, consulta las listas de tipos compatibles en la documentación de Uproot (cajas azules en el texto).
Leer datos desde un archivo#
Abrir el archivo#
Para abrir un archivo para lectura, pasa el nombre del archivo a uproot.open. En los scripts, es una buena práctica usar la instrucción with de Python para cerrar el archivo cuando termines, pero si estás trabajando de forma interactiva, puedes usar una asignación directa.
import skhep_testdata
nombre_del_archivo = skhep_testdata.data_path(
"uproot-Event.root"
) # descarga este archivo de prueba y obtiene una ruta local hacia él
import uproot
archivo = uproot.open(nombre_del_archivo)
Para acceder a un archivo remoto mediante HTTP o XRootD, utiliza una URL que comience con "http://...", "https://...", o "root://...". Si la interfaz de Python para XRootD no está instalada, el mensaje de error explicará cómo instalarla.
Listar contenidos#
Este objeto “archivo” en realidad representa un directorio, y los objetos nombrados en ese directorio son accesibles a través de una interfaz similar a un diccionario. Por lo tanto, keys, values, y items devuelven los nombres de las claves y/o leen los datos. Si solo quieres listar los objetos sin leerlos, utiliza keys. (Esto es similar a ls() de ROOT, excepto que obtienes una lista en Python).
archivo.keys()
['ProcessID0;1', 'htime;1', 'T;1', 'hstat;1']
A menudo, también querrás conocer el tipo de cada objeto, por lo que los objetos uproot.ReadOnlyDirectory también tienen un método classnames, que devuelve un diccionario de nombres de objetos a nombres de clases (sin leerlos).
archivo.classnames()
{'ProcessID0;1': 'TProcessID',
'htime;1': 'TH1F',
'T;1': 'TTree',
'hstat;1': 'TH1F'}
Lectura de un histograma#
Si estás familiarizado con ROOT, TH1F te resultará reconocible como histogramas y TTree como un conjunto de datos. Para leer uno de los histogramas, coloca su nombre entre corchetes:
h = archivo["hstat"]
h
<TH1F (version 2) at 0x7fa978583ec0>
Uproot no realiza ningún tipo de graficación ni manipulación de histogramas, por lo que los métodos más útiles de h comienzan con “to”: to_boost (boost-histogram), to_hist (hist), to_numpy (la tupla de 2 elementos de NumPy que contiene el contenido y los bordes), to_pyroot (PyROOT), etc.
h.to_hist().plot()
[StairsArtists(stairs=<matplotlib.patches.StepPatch object at 0x7fa92a9ed9d0>, errorbar=<ErrorbarContainer object of 3 artists>, legend_artist=<ErrorbarContainer object of 3 artists>)]
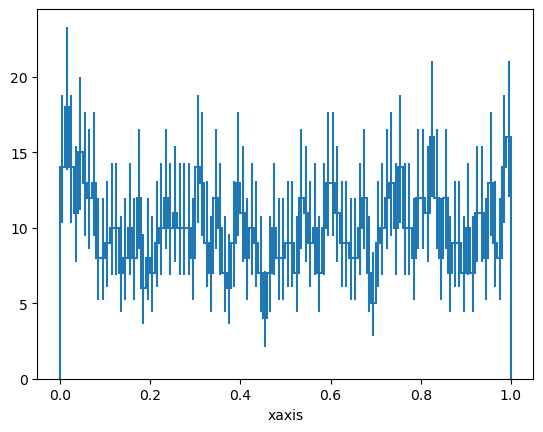
Los histogramas de Uproot también cumplen con el protocolo de graficación UHI, por lo que tienen métodos como values (contenidos de los bins), variances (errores al cuadrado) y axes.
h.values()
array([14., 18., 14., 11., 15., 13., 12., 13., 8., 8., 9., 10., 10.,
7., 8., 10., 8., 12., 6., 8., 7., 9., 10., 12., 10., 11.,
10., 10., 10., 8., 14., 13., 9., 7., 12., 10., 7., 6., 9.,
13., 11., 8., 10., 9., 7., 4., 7., 10., 8., 8., 9., 9.,
7., 12., 11., 9., 10., 7., 10., 13., 13., 11., 9., 9., 8.,
8., 10., 12., 7., 5., 9., 10., 12., 13., 10., 14., 10., 10.,
8., 12., 12., 11., 16., 12., 8., 12., 7., 9., 9., 7., 10.,
7., 11., 11., 8., 13., 9., 8., 14., 16.], dtype=float32)
h.variances()
array([14., 18., 14., 11., 15., 13., 12., 13., 8., 8., 9., 10., 10.,
7., 8., 10., 8., 12., 6., 8., 7., 9., 10., 12., 10., 11.,
10., 10., 10., 8., 14., 13., 9., 7., 12., 10., 7., 6., 9.,
13., 11., 8., 10., 9., 7., 4., 7., 10., 8., 8., 9., 9.,
7., 12., 11., 9., 10., 7., 10., 13., 13., 11., 9., 9., 8.,
8., 10., 12., 7., 5., 9., 10., 12., 13., 10., 14., 10., 10.,
8., 12., 12., 11., 16., 12., 8., 12., 7., 9., 9., 7., 10.,
7., 11., 11., 8., 13., 9., 8., 14., 16.])
list(h.axes[0]) # "x", "y", "z" o 0, 1, 2
[array([0. , 0.01]),
array([0.01, 0.02]),
array([0.02, 0.03]),
array([0.03, 0.04]),
array([0.04, 0.05]),
array([0.05, 0.06]),
array([0.06, 0.07]),
array([0.07, 0.08]),
array([0.08, 0.09]),
array([0.09, 0.1 ]),
array([0.1 , 0.11]),
array([0.11, 0.12]),
array([0.12, 0.13]),
array([0.13, 0.14]),
array([0.14, 0.15]),
array([0.15, 0.16]),
array([0.16, 0.17]),
array([0.17, 0.18]),
array([0.18, 0.19]),
array([0.19, 0.2 ]),
array([0.2 , 0.21]),
array([0.21, 0.22]),
array([0.22, 0.23]),
array([0.23, 0.24]),
array([0.24, 0.25]),
array([0.25, 0.26]),
array([0.26, 0.27]),
array([0.27, 0.28]),
array([0.28, 0.29]),
array([0.29, 0.3 ]),
array([0.3 , 0.31]),
array([0.31, 0.32]),
array([0.32, 0.33]),
array([0.33, 0.34]),
array([0.34, 0.35]),
array([0.35, 0.36]),
array([0.36, 0.37]),
array([0.37, 0.38]),
array([0.38, 0.39]),
array([0.39, 0.4 ]),
array([0.4 , 0.41]),
array([0.41, 0.42]),
array([0.42, 0.43]),
array([0.43, 0.44]),
array([0.44, 0.45]),
array([0.45, 0.46]),
array([0.46, 0.47]),
array([0.47, 0.48]),
array([0.48, 0.49]),
array([0.49, 0.5 ]),
array([0.5 , 0.51]),
array([0.51, 0.52]),
array([0.52, 0.53]),
array([0.53, 0.54]),
array([0.54, 0.55]),
array([0.55, 0.56]),
array([0.56, 0.57]),
array([0.57, 0.58]),
array([0.58, 0.59]),
array([0.59, 0.6 ]),
array([0.6 , 0.61]),
array([0.61, 0.62]),
array([0.62, 0.63]),
array([0.63, 0.64]),
array([0.64, 0.65]),
array([0.65, 0.66]),
array([0.66, 0.67]),
array([0.67, 0.68]),
array([0.68, 0.69]),
array([0.69, 0.7 ]),
array([0.7 , 0.71]),
array([0.71, 0.72]),
array([0.72, 0.73]),
array([0.73, 0.74]),
array([0.74, 0.75]),
array([0.75, 0.76]),
array([0.76, 0.77]),
array([0.77, 0.78]),
array([0.78, 0.79]),
array([0.79, 0.8 ]),
array([0.8 , 0.81]),
array([0.81, 0.82]),
array([0.82, 0.83]),
array([0.83, 0.84]),
array([0.84, 0.85]),
array([0.85, 0.86]),
array([0.86, 0.87]),
array([0.87, 0.88]),
array([0.88, 0.89]),
array([0.89, 0.9 ]),
array([0.9 , 0.91]),
array([0.91, 0.92]),
array([0.92, 0.93]),
array([0.93, 0.94]),
array([0.94, 0.95]),
array([0.95, 0.96]),
array([0.96, 0.97]),
array([0.97, 0.98]),
array([0.98, 0.99]),
array([0.99, 1. ])]
Lectura de un TTree#
Un TTree representa un conjunto de datos potencialmente grande. Obtenerlo del uproot.ReadOnlyDirectory solo devuelve los nombres y tipos de sus TBranch. El método show es una forma conveniente de listar su contenido:
t = archivo["T"]
t.show()
name | typename | interpretation
---------------------+--------------------------+-------------------------------
event | Event | AsGroup(<TBranchElement 'ev...
event/TObject | (group of fUniqueID:u... | AsGroup(<TBranchElement 'TO...
event/TObject/fUn... | uint32_t | AsDtype('>u4')
event/TObject/fBits | uint32_t | AsDtype('>u4')
event/fType[20] | int8_t[20] | AsDtype("('i1', (20,))")
event/fEventName | char* | AsStrings(length_bytes='4')
event/fNtrack | int32_t | AsDtype('>i4')
event/fNseg | int32_t | AsDtype('>i4')
event/fNvertex | uint32_t | AsDtype('>u4')
event/fFlag | uint32_t | AsDtype('>u4')
event/fTemperature | float | AsDtype('>f4', 'float64')
event/fMeasures[10] | int32_t[10] | AsDtype("('>i4', (10,))")
event/fMatrix[4][4] | float[4][4] | AsDtype("('>f4', (4, 4))", ...
event/fClosestDis... | unknown[] | AsObjects(AsArray(False, Tr...
event/fEvtHdr | EventHeader | AsGroup(<TBranchElement 'fE...
event/fEvtHdr/fEv... | int32_t | AsDtype('>i4')
event/fEvtHdr/fEv... | int32_t | AsDtype('>i4')
event/fEvtHdr/fEv... | int32_t | AsDtype('>i4')
event/fTracks | TClonesArray* | AsGroup(<TBranchElement 'fT...
event/fTracks/fTr... | uint32_t[] | AsJagged(AsDtype('>u4'))
event/fTracks/fTr... | uint32_t[] | AsJagged(AsDtype('>u4'))
event/fTracks/fTr... | float[] | AsJagged(AsDtype('>f4'))
event/fTracks/fTr... | float[] | AsJagged(AsDtype('>f4'))
event/fTracks/fTr... | float[] | AsJagged(AsDtype('>f4'))
event/fTracks/fTr... | float[] | AsJagged(AsDtype('>f4'))
event/fTracks/fTr... | Float16_t[] | AsJagged(AsFloat16(0.0, 0.0...
event/fTracks/fTr... | Float16_t[] | AsJagged(AsFloat16(0.0, 0.0...
event/fTracks/fTr... | Float16_t[] | AsJagged(AsFloat16(0.0, 0.0...
event/fTracks/fTr... | float[] | AsJagged(AsDtype('>f4'))
event/fTracks/fTr... | Float16_t[] | AsJagged(AsFloat16(0, 0, 12))
event/fTracks/fTr... | Float16_t[] | AsJagged(AsFloat16(0, 0, 12))
event/fTracks/fTr... | Float16_t[] | AsJagged(AsFloat16(0, 0, 12))
event/fTracks/fTr... | Float16_t[] | AsJagged(AsFloat16(0, 0, 12))
event/fTracks/fTr... | Float16_t[] | AsJagged(AsFloat16(0, 0, 12))
event/fTracks/fTr... | Float16_t[] | AsJagged(AsFloat16(0, 0, 12))
event/fTracks/fTr... | Double32_t[] | AsJagged(AsDouble32(-1.0, 1...
event/fTracks/fTr... | Double32_t[][3] | AsJagged(AsDouble32(-30.0, ...
event/fTracks/fTr... | int32_t[] | AsJagged(AsDtype('>i4'))
event/fTracks/fTr... | int16_t[] | AsJagged(AsDtype('>i2'))
event/fTracks/fTr... | uint32_t[] | AsJagged(AsDtype('>u4'))
event/fTracks/fTr... | unknown[][] | AsObjects(AsArray(True, Fal...
event/fTracks/fTr... | uint32_t[] | AsJagged(AsDtype('>u4'))
event/fTracks/fTr... | uint32_t[] | AsJagged(AsDtype('>u4'))
event/fTracks/fTr... | uint32_t[] | AsJagged(AsDtype('>u4'))
event/fTracks/fTr... | uint32_t[] | AsJagged(AsDtype('>u4'))
event/fTracks/fTr... | uint8_t[][] | AsObjects(AsArray(True, Fal...
event/fTracks/fTr... | float[][3] | AsJagged(AsDtype("('>f4', (...
event/fHighPt | TRefArray* | AsObjects(AsPointer(Model_T...
event/fMuons | TRefArray* | AsObjects(AsPointer(Model_T...
event/fLastTrack | TRef | AsStridedObjects(Model_TRef)
event/fWebHistogram | TRef | AsStridedObjects(Model_TRef)
event/fH | TH1F | AsObjects(Model_TH1F)
event/fTriggerBits | TBits | AsGroup(<TBranchElement 'fT...
event/fTriggerBit... | (group of fTriggerBit... | AsGroup(<TBranchElement 'fT...
event/fTriggerBit... | uint32_t | AsDtype('>u4')
event/fTriggerBit... | uint32_t | AsDtype('>u4')
event/fTriggerBit... | uint32_t | AsDtype('>u4')
event/fTriggerBit... | uint32_t | AsDtype('>u4')
event/fTriggerBit... | uint8_t[] | AsJagged(AsDtype('uint8'), ...
event/fIsValid | bool | AsDtype('bool')
Ten en cuenta que puedes obtener la misma información de keys (un uproot.TTree es similar a un diccionario), typename e interpretation.
t.keys()
['event',
'event/TObject',
'event/TObject/fUniqueID',
'event/TObject/fBits',
'event/fType[20]',
'event/fEventName',
'event/fNtrack',
'event/fNseg',
'event/fNvertex',
'event/fFlag',
'event/fTemperature',
'event/fMeasures[10]',
'event/fMatrix[4][4]',
'event/fClosestDistance',
'event/fEvtHdr',
'event/fEvtHdr/fEvtHdr.fEvtNum',
'event/fEvtHdr/fEvtHdr.fRun',
'event/fEvtHdr/fEvtHdr.fDate',
'event/fTracks',
'event/fTracks/fTracks.fUniqueID',
'event/fTracks/fTracks.fBits',
'event/fTracks/fTracks.fPx',
'event/fTracks/fTracks.fPy',
'event/fTracks/fTracks.fPz',
'event/fTracks/fTracks.fRandom',
'event/fTracks/fTracks.fMass2',
'event/fTracks/fTracks.fBx',
'event/fTracks/fTracks.fBy',
'event/fTracks/fTracks.fMeanCharge',
'event/fTracks/fTracks.fXfirst',
'event/fTracks/fTracks.fXlast',
'event/fTracks/fTracks.fYfirst',
'event/fTracks/fTracks.fYlast',
'event/fTracks/fTracks.fZfirst',
'event/fTracks/fTracks.fZlast',
'event/fTracks/fTracks.fCharge',
'event/fTracks/fTracks.fVertex[3]',
'event/fTracks/fTracks.fNpoint',
'event/fTracks/fTracks.fValid',
'event/fTracks/fTracks.fNsp',
'event/fTracks/fTracks.fPointValue',
'event/fTracks/fTracks.fTriggerBits.fUniqueID',
'event/fTracks/fTracks.fTriggerBits.fBits',
'event/fTracks/fTracks.fTriggerBits.fNbits',
'event/fTracks/fTracks.fTriggerBits.fNbytes',
'event/fTracks/fTracks.fTriggerBits.fAllBits',
'event/fTracks/fTracks.fTArray[3]',
'event/fHighPt',
'event/fMuons',
'event/fLastTrack',
'event/fWebHistogram',
'event/fH',
'event/fTriggerBits',
'event/fTriggerBits/fTriggerBits.TObject',
'event/fTriggerBits/fTriggerBits.TObject/fTriggerBits.fUniqueID',
'event/fTriggerBits/fTriggerBits.TObject/fTriggerBits.fBits',
'event/fTriggerBits/fTriggerBits.fNbits',
'event/fTriggerBits/fTriggerBits.fNbytes',
'event/fTriggerBits/fTriggerBits.fAllBits',
'event/fIsValid']
t["event/fNtrack"]
<TBranchElement 'fNtrack' at 0x7fa91b2b62a0>
t["event/fNtrack"].typename
'int32_t'
t["event/fNtrack"].interpretation
AsDtype('>i4')
(Si un uproot.TBranch no tiene interpretation, no se puede leer con Uproot.)
La forma más directa de leer datos de un uproot.TBranch es llamando a su método array.
t["event/fNtrack"].array()
[600, 604, 603, 594, 595, 598, 595, 600, 592, 598, ..., 603, 605, 609, 599, 587, 598, 600, 596, 593] ------------------ type: 1000 * int32
Consideraremos otros métodos en la próxima lección.
Leyendo un… ¿qué es eso?#
Este archivo también contiene una instancia del tipo TProcessID. Estos tipos de objetos no son típicamente útiles en el análisis de datos, pero Uproot logra leerlo de todos modos porque sigue ciertas convenciones (tiene “streamers de clase”). Se presenta como un objeto genérico con una propiedad all_members para sus miembros de datos (a través de todas las superclases).
archivo["ProcessID0"]
<TProcessID (version 1) at 0x7fa91b8c9580>
archivo["ProcessID0"].all_members
{'@fUniqueID': 0,
'@fBits': 50331648,
'fName': 'ProcessID0',
'fTitle': '3ec87674-3aa2-11e9-bb02-0301a8c0beef'}
Aquí hay un ejemplo más útil de eso: una búsqueda de supernovas con el experimento IceCube tiene clases personalizadas para sus datos, que Uproot lee y representa como objetos con all_members.
icecube = uproot.open(skhep_testdata.data_path("uproot-issue283.root"))
icecube.classnames()
{'config;1': 'TDirectory',
'config/analysis;1': 'SN_Analysis_Configuration_t',
'config/detector;1': 'I3Eval_t',
'config/run;1': 'SN_File_t',
'sn_all;1': 'TTree',
'sn_gps;1': 'TTree',
'sn_range;1': 'TTree',
'sn_o2rout;1': 'TTree',
'sn_o2cand;1': 'TTree',
'sn_omwatch;1': 'TTree',
'sn_sigsim;1': 'TTree'}
icecube["config/detector"].all_members
{'@fUniqueID': 0,
'@fBits': 50331648,
'theDataArray': <Sni3DataArray (version 1) at 0x7fa91999fdd0>,
'NumberOfChannels': 5160,
'NoAvailableSlices': -1,
'AvailableDataSize': 0,
'mGPSCardId': 0,
'mGPSPrescale': 20000000,
'mGPSEventNo': 92824,
'mScalerCardId': 0,
'mScalerStartChannel': 0,
'StartUTC': 272924620173109013,
'MaxChannels': 5160,
'mMaxJitterLogs': 20,
'Channel': <I3Eval_t::ChannelContainer_t (version 1) at 0x7fa9704d20c0>,
'ChannelIDMap': <STLMap {46612627560: 896, ..., 281410180683757: 2689} at 0x7fa91b444650>,
'BadChannelIDSet': <STLSet {58348614635591, 60068372029697, ..., 258905191174588} at 0x7fa91999ffe0>,
'ChannelID': array([ 47303335284587, 20579555797555, 106634453247646, ...,
255380957221937, 107432791511293, 280205879548048],
shape=(5160,), dtype='>i8'),
'Deadtime': array([250., 250., 250., ..., 250., 250., 250.],
shape=(5160,), dtype='>f8'),
'Efficiency': array([1. , 1. , 1. , ..., 1.35, 1.35, 1.35],
shape=(5160,), dtype='>f8')}
icecube["config/detector"].all_members["ChannelIDMap"]
<STLMap {46612627560: 896, ..., 281410180683757: 2689} at 0x7fa91b444650>
Escribiendo datos en un archivo#
La capacidad de Uproot para escribir datos es más limitada que su capacidad para leer datos, pero algunos casos útiles son posibles.
Abrir archivos para escribir#
Primero que nada, un archivo debe ser abierto para escribir, ya sea creando un archivo completamente nuevo o actualizando uno existente.
archivo_nuevo = uproot.recreate("archivo-completamente-nuevo.root")
archivo_existente = uproot.update("archivo-existente.root")
(Uproot no puede escribir a través de una red; los archivos de salida deben ser locales.)
Escribiendo cadenas y histogramas#
Estos objetos uproot.WritableDirectory tienen una interfaz similar a un diccionario: puedes poner datos en ellos asignando a corchetes.
archivo_nuevo["una_cadena"] = "Este objeto va a ser un TObjString."
archivo_nuevo["un_histograma"] = archivo["hstat"]
import numpy as np
archivo_nuevo["un_directorio/otro_histograma"] = np.histogram(
np.random.normal(0, 1, 1000000)
)
En ROOT, el nombre de un objeto es una propiedad del objeto, pero en Uproot, es una clave en el TDirectory que contiene el objeto, por lo que el nombre está en el lado izquierdo de la asignación, entre corchetes. Solo se admiten los tipos de datos enumerados en el cuadro azul en la documentación: principalmente solo histogramas.
Escribiendo TTrees#
Los TTrees son potencialmente grandes y pueden no caber en la memoria. Generalmente, necesitarás escribirlos en lotes.
Una forma de hacer esto es asignar el primer lote y extend con lotes posteriores:
import numpy as np
archivo_nuevo["tree1"] = {
"x": np.random.randint(0, 10, 1000000),
"y": np.random.normal(0, 1, 1000000),
}
archivo_nuevo["tree1"].extend(
{"x": np.random.randint(0, 10, 1000000), "y": np.random.normal(0, 1, 1000000)}
)
archivo_nuevo["tree1"].extend(
{"x": np.random.randint(0, 10, 1000000), "y": np.random.normal(0, 1, 1000000)}
)
Otra forma es crear un TTree vacío con uproot.WritableDirectory.mktree, de modo que cada escritura sea una extensión.
archivo_nuevo.mktree("tree2", {"x": np.int32, "y": np.float64})
archivo_nuevo["tree2"].extend(
{"x": np.random.randint(0, 10, 1000000), "y": np.random.normal(0, 1, 1000000)}
)
archivo_nuevo["tree2"].extend(
{"x": np.random.randint(0, 10, 1000000), "y": np.random.normal(0, 1, 1000000)}
)
archivo_nuevo["tree2"].extend(
{"x": np.random.randint(0, 10, 1000000), "y": np.random.normal(0, 1, 1000000)}
)
Se dan consejos de rendimiento en la próxima lección, pero en general, es más beneficioso escribir pocos lotes grandes en lugar de muchos lotes pequeños.
Los únicos tipos de datos que se pueden asignar o pasar a extend están listados en la caja azul en esta documentación. Esto incluye arrays dentados (descritos en la lección después de la próxima), pero no tipos más complejos.
